A IA RESOLVE CASE POR COMPLETO UN DOS GRANDES ENIGMAS DA BIOLOXÍA
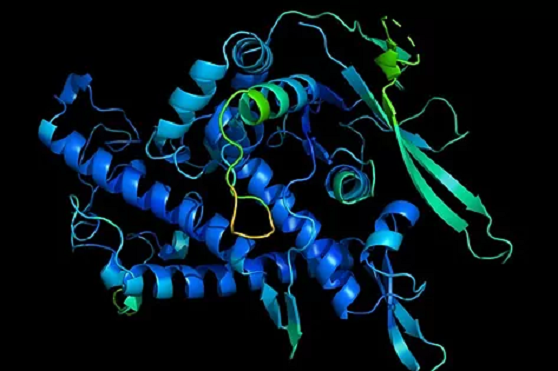
Estrutura tridimensional dunha proteína /
As proteínas son esenciais para a vida. Son moléculas complexas compostas por cadeas de aminoácidos e as funcións que desempeñan dependen en gran medida da súa estrutura en tres dimensións, que é única para cada proteína. Por iso, pescudar como adquiren a súa forma tridimensional foi un dos anhelos dos biólogos desde hai 50 anos. Un enigma que se coñece como o problema do pregamento da proteína (protein folding problem) e que agora a intelixencia artificial parece haber resolvido case totalmente.
Así o asegurou o luns compañía DeepMind. A través do deep learning (aprendizaxe profunda), a última versión do seu sistema de intelixencia artificial AlphaFold conseguiu descifrar como as proteínas adquiren a súa forma, tal e como recoñeceron os organizadores do concurso CASP (Avaliación crítica das técnicas para a predición estrutural proteica, Critical Assessment of protein Structure Prediction), un experimento comunitario que se celebra dúas veces ao ano.
"Este fito demostra o impacto que a intelixencia artificial pode ter nos descubrimentos científicos e o seu potencial para acelerar o progreso nalgúns dos campos máis importantes que explican e modelan o noso mundo", sinalou a compañía DeepMind, fundada en 2010 e adquirida por Google hai seis anos.
As proteínas xogan un papel esencial en moitos procesos, desde o desenvolvemento dunha enfermidade e por tanto no descubrimento de tratamentos médicos para combatelas, á procura de encimas que destrúan refugallos industriais. Por iso, coñecer como adquiren a súa forma, sinalan os enxeñeiros de DeepMind, pode axudar a acelerar o desenvolvemento de fármacos para tratar enfermidades, incluíndo a Covid-19, e a mellorar procesos industriais. Pero tamén a coñecer mellor como funciona o corpo humano e o mundo.
"Isto vai cambiar a medicina, vai cambiar a investigación e a bioinxeñaría. Vai cambiar todo", asegura Andrei Lupas, biólogo no Instituto Max Planck de Bioloxía do Desenvolvemento en Tübingen, Germany. Tal e como declarou á revista Nature, o programa AlphaFold axudoulle xa a descubrir a estrutura dunha proteína no seu laboratorio coa que traballaba desde hai unha década.
A función biolóxica que desempeña unha proteína depende do seu correcto pregamento. Trátase dun proceso espontáneo e termodinámicamente irreversible: se unha proteína non se prega correctamente, non será funcional e, por tanto, non poderá cumprir coa súa función biolóxica.
O problema do dobramento da proteína xurdiu nos anos 60, coa aparición das primeiras estruturas de proteínas con resolución atómica. Algunhas tiñan unha forma interna esperada pero outras non. Así que se expuxo este enigma, que á súa vez encerra tres quebracabezas relacionados para pescudar tres cuestiones: que é o código de dobramento, cal é o mecanismo de dobramento e se se pode predicir a estrutura orixinal dunha proteína a partir da súa secuencia de aminoácidos.
Nos últimos anos houbo avances para resolver este enigma e na actualidade as estruturas de pequenas proteínas pódense predicir con varias técnicas complexas de análises de materiais como a criomicroscopía electrónica, a resonancia magnética nuclear ou a cristalografía de raios-X. Trátase de métodos complexos que requiren longos e laboriosos experimentos e equipos tecnolóxicos caros para levalos a cabo.
O programa de DeepMind aprendeu a recoñecer como se forman estas estruturas e é capaz de pescudar a forma tridimensional de proteína en cuestión de horas.
"Trátase de algo moi importante. En certo xeito, o problema foi resolto", declarou John Moult, biólogo computacional na Unversidad de Maryland e un dos fundadores do concurso CASP para mellorar os métodos computacionais que permiten predicir de forma correcta as estruturas das proteínas.
Tal e como explicaron os enxeñeiros de DeepMind, o seu programa AlphaFold foi adestrado cunha base de datos pública que contén 170.000 estruturas de proteínas, a partir das cales aprendeu a identificar a súa forma tridimensional. A primeira versión do programa de intelixencia artificial AlphaFold participou no concurso CASP en 2018, logrando predicir a estrutura das proteínas cunha fiabilidade de 60 (nunha escala de 100 puntos). Na edición celebrada agora, a nova versión de AlphaFold logrou unha puntuación de 90 sobre 100.
Os enxeñeiros de DeepMind esperan que o seu programa axúdelles a identificar proteínas que funcionaron de maneira incorrecta e a pescudar por que, o que podería axudar a desenvolver de forma máis precisa e máis rápida novos fármacos. A IA complementaría aos métodos experimentais actuais para o desenvolvemento de medicamentos.
A predición da estrutura dunha proteína, engaden, podería ser útil tamén para responder diante futuras pandemias como a do coronavirus. No principio do ano, foron capaces de predicir as estruturas de varias proteínas do coronavirus SARS- CoV-2, entre elas a ORF3a e a ORF8.
FONTE: Teresa Guerrero/elmundo.es/ciencia
0 comentarios